Monográfico. Clasificación con SVM en R
Las máquinas de vectores de soporte, Support Vector Machines, SVM a partir de ahora, son un conjunto de técnicas estadísticas que nos permiten clasificar una población en función de la partición en subespacios de múltiples variables. Parte de la idea de dividir de forma lineal un conjunto de múltiples dimensiones. Creamos muchos hiperplanos que nos dividen las observaciones. Es una técnica que está ganando popularidad y que por supuesto podemos realizarla con R. Para ello tenemos algunos paquetes específicos como kvm, svmlight y el e1071. Este último es al que pretendo acercarme hoy.
El SVM es un algoritmo que, a partir del producto escalar de vos vectores multidimensionales, busca hiperplanos que separen los grupos. La función que define este producto escalar la denominaremos kernel y puede ser lineal, polinómica, radial o sigmoidal. Para clasificación el SVM se plantea como un problema de programación lineal en el que buscamos maximizar la distancia entre categorías sujeto a un coste y a un número óptimo de patrones de entrenamiento. Para entender mejor su funcionamiento trabajamos un ejemplo bidimensional:
#Simulación de un conjunto de datos bivariante
x=c(rnorm(500,1000,100),rnorm(500,2000,200),rnorm(500,3000,400))
y=c(abs(rnorm(500,50,25)),rnorm(500,200,50),rnorm(500,100,30))
grupo=as.factor(c(rep(1,500),rep(2,500),rep(3,500)))
datos=data.frame(x,y,grupo)
Tenemos un data frame con 3 variables, la variable grupo nos define el grupo clasificador. Gráficamente:
#Gráfico sin modelo
require(lattice);
xyplot(y~x,group=grupo,data=datos,cex=1,pch=13)
Los 3 grupos quedan claramente diferenciados, aunque se busca un cierto solape entre 2 de ellos. Creamos un conjunto de entrenamiento y una muestra de validación para comprobar posteriormente su funcionamiento:
#Creamos una muestra para entrenar el modelo
elimina=sample(1:nrow(datos),300)
muestra=datos[elimina,]
entrena=datos[-elimina,]
#Realización de modelo con la librería e1071
library(e1071);
modelo=svm(grupo~y+x,data=entrena,method="C-classification",
kernel="radial",cost=10,gamma=.1)
Entrenamos el modelo con 1.200 observaciones. El método empleado es el C-classification para clasificar nuestros registros, esto requiere que la variable dependiente sea un factor. La función de núcleo es radial, el coste es una medida que nos limita el error, a mayor coste mayor tiempo de computación y gamma es una medida necesaria para la función kernel excepto en el caso lineal. Veamos el comportamiento del modelo con la muestra de 300 registros que hemos dejado:
#Analizamos el comportamiento
predic=data.frame(predict(modelo,muestra))
muestra=cbind(muestra,predic=predic)
table(muestragrupo,muestrapredict.modelo..muestra.)
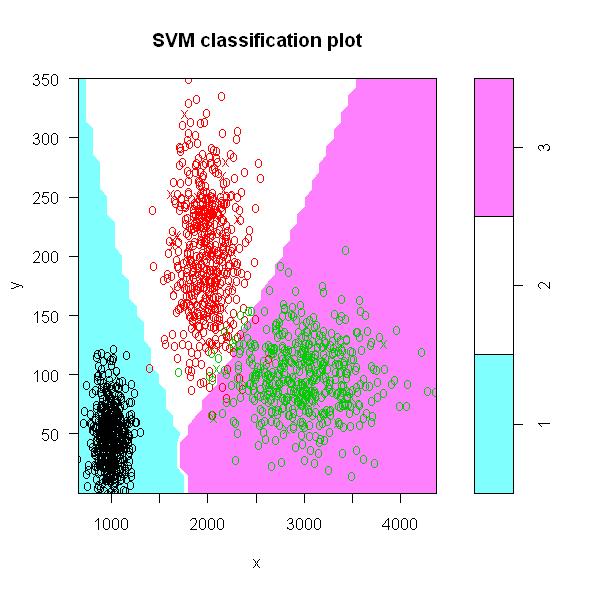
Obtenemos un buen comportamiento predictor. Para el caso bivariante podemos estudiar el comportamiento de forma gráfica:
#Análisis gráfico
win.graph();plot(modelo,datos)
Espero que despierte vuestra curiosidad con esta técnica y para aquellos que la conocen comentadnos que paquete de R empleáis o que software recomendáis. Por otro lado si alguien ha comparado las SVM con las redes neuronales que nos lo cuente porque son técnicas con muchas similitudes pero con una ventaja clara para las SVM, la facilidad de comprensión.
Saludos.